studio concluso
Antonio Francavilla, Italian Institute for Genomic Medicine, Torino
Borsa di Studio triennale
Studio sulla Celiachia
Area: Genetica
Tutor (Capo Laboratorio): Prof. Paolo Vineis, Italian Institute for Genomic Medicine, Torino
Scheda dello Studio
- Numero del Finanziamento (Grant): FC 015/2017
- Titolo: Studio dei microRNA e del microbiota intestinale in feci di soggetti adulti affetti da malattia Celiaca.
- Area Scientifica: Genetica
- Durata: Progetto Triennale
- Ricercatore Titolare: Dott. Antonio Francavilla, Italian Institute for Genomic Medicine (IIGM), Torino
- Tutor (Capo Laboratorio): Prof. Paolo Vineis, Italian Institute for Genomic Medicine, Torino
- Collaborazioni: Dott. Davide Giuseppe Ribaldone e Dott. Gian Paolo Caviglia, Unità di Gastroenterologia, Dipartimento di Scienze Mediche, Torino; Prof. Mauro Bruno, Unità di Gastroenterologia ed Endoscopia Digestiva, “Città della Salute e della Scienza” Torino
Pubblicazioni derivanti dal Grant:
- Francavilla A, Gagliardi A, Piaggeschi G, Tarallo S, Cordero F, Pensa RG, Impeduglia A, Caviglia GP, Ribaldone DG, Gallo G, Grioni S, Ferrero G, Pardini B, Naccarati A. Faecal miRNA profiles associated with age, sex, BMI, and lifestyle habits in healthy individuals. Sci Rep. 2021 Oct 19;11(1):20645. doi: 10.1038/s41598-021-00014-1. PMID: 34667192; PMCID: PMC8526833. https://pubmed.ncbi.nlm.nih.gov/34667192/
- Francavilla A, Ferrero G, Pardini B, Tarallo S, Zanatto L, Caviglia GP, Sieri S, Grioni S, Francescato G, Stalla F, Guiotto C, Crocella L, Astegiano M, Bruno M, Calvo PL, Vineis P, Ribaldone DG, Naccarati A. Gluten-free diet affects fecal small non-coding RNA profiles and microbiome composition in celiac disease supporting a host-gut microbiota crosstalk. Gut Microbes. 2023 Jan-Dec;15(1):2172955. doi: 10.1080/19490976.2023.2172955. PMID: 36751856; PMCID: PMC9928459. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC9928459/
Lo studio
Cosa si è voluto studiare e perché?
La malattia celiaca (CD) è sempre più diffusa e si cura solo aderendo ad una dieta senza glutine (GFD). Pertanto, nuovi biomarcatori per la diagnosi di CD e per il monitoraggio dell’aderenza alla GFD o i suoi effetti ne migliorerebbero la gestione clinica. In questo ambito troviamo i piccoli RNA non codificanti (sncRNA), tra i quali i più famosi sono i miRNA, ovvero molecole che regolano l’espressione dei geni. Recentemente, è stata associata l’assunzione di nutrienti specifici con l’espressione di alcuni miRNA in individui sani e a questo proposito, lo studio di queste molecole in relazione alla GFD potrebbe evidenziare l’impatto di questa dieta sui celiaci nel corso degli anni. Inoltre, è noto che la dieta influenza il microbiota intestinale e varie evidenze mostrano l’associazione tra disbiosi e malattie gastrointestinali, celiachia inclusa. Più recentemente è stato mostrato come i miRNA fecali regolino direttamente l’espressione di specifici geni batterici essenziali per il mantenimento del normale microbiota e viceversa. Ciò suggerisce che l’asse dieta-microbiota-miRNA potrebbe essere cruciale per la regolazione dell’espressione genica dell’ospite. Tuttavia, il rapporto tra miRNA e microbioma in relazione alla GFD è ancora ignoto. Con questi presupposti, l’obiettivo dello studio è stato analizzare l’espressione di miRNA e altri sncRNA, e la composizione del microbiota nelle feci i) in celiaci in dieta aglutinata sia risultati positivi al test di anticorpi anti-transglutaminasi, con sintomi gastrointestinali e/o aderenti alla GFD da poco (tCD-TG+), sia invece in rigorosa GFD e risultati negativi al test serologico, tCD) rispetto a dei controlli sani senza alcuna restrizione nella dieta; ii) in relazione agli anni di aderenza alla GFD; e infine iii) integrando i dati di miRNA, microbioma e nutrienti per identificare i migliori biomarcatori per distinguere differenti categorie di dai controlli sani.
La Metodologia
In totale, sono stati arruolati 127 individui (pazienti celiaci e soggetti sani onnivori e senza specifiche intolleranze alimentari con una pari distribuzione di maschi e femmine e di medesima età). Per ognuno sono stati raccolti un campione di plasma, siero e feci oltre a due questionari (uno sulle abitudini alimentari e uno sugli stili di vita). Fatta eccezione per i soggetti già clinicamente testati , in tutti gli altri partecipanti che hanno fornito un campione di sangue sono stati misurati i livelli di anticorpi anti-transglutaminasi.
I livelli di espressione di miRNA e altri sncRNA sono stati valutati in plasma e feci mediante next-generation sequencing (small RNA-sequencing). Allo stesso modo, la composizione del microbioma intestinale è stata analizzata in campioni di feci mediante una tecnica di metagenomica avanzata (shotgun metagenomic sequencing). I dati generati da entrambe le tecniche di sequenziamento sono stati analizzati con i più avanzati approcci bioinformatici.
Quali Risultati e Quali Conclusioni?
Diversi miRNA hanno riportato specifici profili di espressione nelle feci dei tCD, e in particolare dei tCD-TG+. Analisi funzionali hanno evidenziato che i miRNA più espressi nei tCD-TG+ sono coinvolti in vari processi biologici legati ad uno stato infiammatorio, coerentemente con la condizione di questo gruppo di soggetti. E’ interessante che i livelli di espressione di alcuni di questi miRNA (miR-139-3p, miR-641-3p, miR-4279, miR-4533, miR-6505-3p e miR-6720-3p) sono risultati variare in base anche agli anni di aderenza alla GFD, indipendentemente dall’età dei soggetti. Inoltre, anche altre classi di sncRNA, principalmente piRNA e tRNA, sono risultate alterate in campioni fecali solo nel gruppo dei tCD-TG+ rispetto ai controlli, dimostrando un probabile loro coinvolgimento in processi infiammatori. In campioni di plasma degli stessi soggetti invece non sono state osservate alcune differentze nei livelli di espressione dei miRNA e degli altri sncRNA.
L’analisi del microbioma intestinale ha evidenziato alcune differenze fra i gruppi analizzati. A livello di specie, è stata osservata una riduzione nei livelli di abbondanza di Bifidobacterium longum, Roseburia sp CAG 309, Ruminococcus bicirculans, Ruminococcus callidus ed Eubacterium sp CAG 274 nei tCD rispetto ai controlli, mentre Roseburia inulinivorans è risultata più abbondante. Nel gruppo tCD-TG+, quattro specie microbiche erano più abbondanti (Veillonella atypica, Veillonella tobetsuensis, Streptococcus sanguinis e Haemophilus parainfluenzae) mentre Firmicutes CAG 83 e Ruminococcus bicirculans erano meno abbondanti rispetto ai controlli sani. In generale, il profilo funzionale metabolico microbico dei celiaci ha rivelato una minore abbondanza di geni coinvolti nel metabolismo dell’amido. Questo risultato potrebbe essere correlato alle differenze tra soggetti CD e controlli dovute all’adesione alla GFD che include prodotti senza glutine caratterizzati da un diverso contenuto di amido e carboidrati.
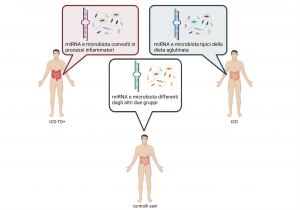
Figura 1 Differenti profili di miRNA e diversa composizione del microbiota intestinale in soggetti celiaci non aderenti a rigorosa GFD o in dieta da poco tempo (tCD-TG+), in soggetti celiaci in rigorosa GFD (tCD) e in controlli sani (healthy).
In ultima analisi, integrando i dati di miRNA, del microbioma e dei dati dei nutrienti assunti giornalmente e sottoponendoli ad analisi con un classificatore basato su tecniche di machine learning si è visto che un pannello basato sui suddetti dati permette di discriminare accuratamente i soggetti celiaci dai controlli. Questi risultati mostrano che l’integrazione di questi dati diversi e in particolare quelli riguardanti i miRNA fecali e il microbioma intestinale, potrebbero fornire uno strumento diagnostico e di monitoraggio della celiachia.
Quali prospettive e quali benefici per i pazienti celiaci?
Il nostro studio presenta la prima caratterizzazione su larga scala dei miRNA fecali e della composizione microbica intestinale in relazione alla Celiachia e alla dieta senza glutine con tecnologie di biologia molecolare e bionformatica all’avanguardia. I risultati mostrano la modulazione della composizione del miRNoma umano e del microbioma intestinale come conseguenza alla dieta aglutinata e dello stato infiammatorio nei soggetti ancora sintomatici e non aderenti alla dieta aglutinata in maniera scrupolosa. I nostri dati forniscono anche nuove evidenze per migliorare la nostra comprensione del ruolo dei piccoli RNA nel crosstalk ospite-microbioma in condizioni fisiologiche e patologiche. Inoltre l’analisi integrata dei miRNA e del microbioma intestinale può fornire spunti per l’ideazione di modelli dietetici personalizzati più accurati e raccomandazioni nel trattamento e nel monitoraggio della celiachia in maniera non invasiva.
Eventuali sviluppi futuri del progetto
Aumentare il numero di soggetti analizzati in questo studio è sicuramente una prospettiva futura importante per aumentare la rilevanza dei risultati ottenuti. A questo proposito, studiare i miRNA e il microbioma intestinale anche in soggetti sani ma aderenti ad una dieta aglutinata, fornirebbe un quadro più completo sull’influenza che questa dieta esercita sui profili delle suddette molecole e della flora batterica intestinale. Inoltre, considerati gli ottimi risultati ottenuti circa la possibilità di utilizzare un pannello di miRNA e specie batteriche per discriminare fra due diversi gruppi di celiaci in dieta, estendere lo studio anche in soggetti con celiachia attiva e quindi non ancora aderenti alla dieta aglutinata, potrebbe aiutare a capire il potere di questi pannelli anche in ambito diagnostico ed eventualmente costituire una speranza di miglioramento degli attuali test diagnostici invasivi.
Legenda
miRNA: diminutivo di microRNA, classe di molecole a RNA di piccole dimensioni coinvolte nel processo di regolazione dell’espressione genica. La loro stabilità e facile reperibilità nei fluidi biologici ha portato ad un grande interesse nell’investigare la loro possibile applicazione come biomarcatori non invasivi in diverse malattie tra cui anche quelle gastrointestinali.
sncRNA: classe di molecole a RNA di piccola taglia (18-200 nt) che include diverse specie fra cui i miRNA.
Microbiota: composizione microbica tipica di varie parti del nostro organismo, che include varie specie batteriche ed in misura minore archeobatteri, virus e miceti che ci abitano. L’aggettivo intestinale serve a chiarire che si tratta di un microbiota specifico di questo tratto e quindi diverso da quelli presenti in altri organi del nostro corpo o altri habitat naturali.
Microbioma: caratterizzazione dei microbi a livello genico, genoma microbico.
Disbiosi: alterazioni del microbiota tali da indurre variazioni nella normale funzionalità intestinale
Sequenziamento: tecnica di lettura della sequenza degli acidi nucleici (DNA/RNA).
Tecniche Metagenomiche: tecniche molecolari di indagine del microbiota, ed in particolare al sequenziamento completo di tutto il genoma microbico.
Machine Learning: forma di intelligenza artificiale che effettua previsioni di dati sulla base di dati forniti in input.
Biomarcatore: qualsiasi molecola o fattore biologico in grado di individuare una specifica condizione.
Alterazioni epigenetiche: variazioni genetiche che non interessano la sequenza del DNA
